« Voir pour mieux comprendre », telle est la devise de la biologie structurale moderne. Un domaine qui vise à obtenir la structure des objets biologiques pour mieux comprendre le rôle d’une molécule donnée dans la cellule. La biologie structurale moderne est vraisemblablement née dès 1953 avec la publication de la structure de la double hélice de l’ADN par Watson et Crick 1 et quelques années plus tard avec les premières structures de protéines par Kendrew et Perutz. Depuis, la biologie structurale a fourni aux chercheurs de précieuses informations pour comprendre le vivant et le soigner.
Une histoire technique
Les techniques disponibles ont émergé et évolué chacune à leur tour : la cristallographie aux rayons X, déjà maitrisée dans les années 1910 a connu une révolution dans les années 1990 grâce, entre autres, à l’utilisation de la cryocongélation de cristaux et à la qualité des synchrotrons qui génèrent des rayons X. La résonance magnétique nucléaire, découverte par hasard dans les années 1940 a émergé en biologie structurale dans les années 2000 et est encore utilisée aujourd’hui, surtout pour étudier la dynamique des protéines. La microscopie électronique, née dans les années 1930 a longtemps été considérée, en biologie, comme une paire de jumelles pour voir l’infiniment petit mais sans donner d’informations structurales à haute résolution.
Le problème : les molécules du vivant sont minuscules. Par exemple, la protéine Spike du SARS-CoV‑2 pèse ~180–200 kDa2 — l’équivalent de trois molécules d’hémoglobine3. Les images obtenues par microscopie électronique sont très bruitées, en raison de l’interaction du faisceau d’électrons du microscope avec l’échantillon. Résultat : on ne distingue plus rien sur les images si l’objet que l’on observe est trop petit ! Ce n’est que depuis une dizaine d’années qu’une révolution a pu voir le jour : la conception des microscopes a été améliorée, la qualité et la rapidité des détecteurs utilisés permettent d’enregistrer des images de meilleure qualité, les logiciels d’aide au traitement des données sont plus intuitifs, plus performants et la vitesse de certains calculs a pu être améliorée d’un facteur 50 grâce aux cartes graphiques des ordinateurs. Si la pandémie du SARS-CoV‑2 était apparue il y a une quinzaine d’années, nous n’aurions jamais été en mesure d’obtenir sa structure aussi rapidement et aussi précisément pour lutter contre elle !
Mieux et plus vite
Née en Allemagne, la microscopie électronique a d’abord servi la physique. La biologie a bénéficié des développements faits en physique et les premières images d’échantillons biologiques datent des années 1950. Ce n’est qu’en 1968 que les américains de Rosier et Klug démontrent qu’à partir d’images 2D prises avec un microscope électronique permettent de remonter à la structure 3D de l’objet étudié. La première structure d’une protéine membranaire provient d’une bactérie. Elle date de 1975 et a été réalisée par les pionniers britanniques Unwin et Henderson. (Voir photo ci-contre). Henderson a reçu un prix Nobel en 2017 pour ses travaux pionniers pendant plus de 40 ans avec les chercheurs Frank et Dubochet.

Il va falloir attendre 40 ans de plus pour obtenir la résolution quasi-atomique (3Å de résolution moyenne) du ribosome humain (fait de plusieurs brins d’ARN et d’une cinquantaine de protéines). Aujourd’hui, c’est plus de 10 structures par jour qui sont déposées dans les bases de données des chercheurs et plus de 20% d’entre elles sont des données à haute résolution.
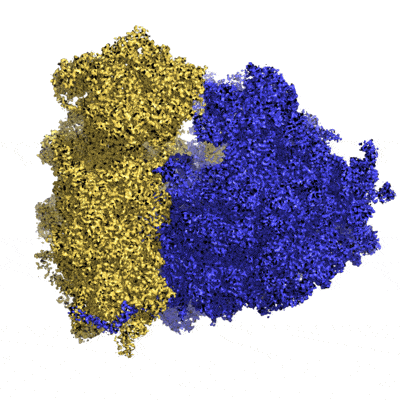
Dès janvier 2022, la structure précise de la protéine Spike du variant omicron SARS-CoV‑2 (apparu seulement en novembre 2021) et de sa cible humaine (le récepteur pulmonaire ACE2) a été révélée. Ceci a permis de mettre en place de nouvelles stratégies pour lutter plus efficacement contre l’infection de ce virus dans nos poumons6. Aussi, la connaissance de la disposition exacte des fibres amyloïdes impliquées dans la maladie d’Alzheimer a permis de concevoir des médicaments ralentissant l’accumulation de ces fibres dans notre cerveau7. Ainsi, de plus en plus de modèles biologiques sont revisités et améliorés rapidement grâce aux structures 3D à haute résolution obtenues avec les dernières avancées des microscopes électroniques. Des verrous technologiques sont en train de s’ouvrir pour maintenant observer des protéines seules ou en complexe avec d’autres partenaires afin de mieux comprendre leur rôle dans la cellule. Pas étonnant que l’industrie pharmaceutique investisse massivement dans cette technique pour cribler plus facilement et rapidement leurs nouvelles molécules thérapeutiques !
Les limites de la technique
Un microscope électronique fonctionne sous ultravide (10-8 mbar) afin que les électrons qui y circulent génèrent le moins de bruit parasite possible. Le seul moyen d’observer un échantillon biologique est de le transformer en glaçon solide (par vitrification) pour que l’échantillon, une fois dans le microscope, reste sous forme solide tout en gardant sa forme native : c’est ce qu’on appelle la cryo-microscopie électronique (ou cryo-EM en anglais) !

Le support sur lequel l’échantillon est déposé est, en général, une membrane de carbone perforée de 10 nm d’épaisseur qui repose sur une charpente de cuivre formant des carreaux de 100x100 µm. L’échantillon va être piégé dans les trous de la membrane perforée et l’étape la plus critique va être de former une fine pellicule dans chaque trou. La charpente représente un disque de 3 mm de diamètre (appelé grille). Cette grille est ensuite déposée dans le microscope électronique.
Le faisceau d’électrons utilisé (généré par le microscope électronique) va traverser l’échantillon déposé sur la grille. Les images enregistrées par transmission vont renseigner sur l’organisation 3D des atomes que le faisceau rencontre, mais il génère aussi beaucoup de bruits parasites. C’est pourquoi il est nécessaire d’enregistrer des milliers de clichés pour moyenner les informations et obtenir un bon rapport signal sur bruit, afin de déterminer sans ambiguïté la forme des objets biologiques étudiés.
Sur chaque cliché, on pourra alors observer quelques dizaines d’objets biologiques (appelés particules). Les vues observées sur les clichés correspondent à des projections différentes d’un même objet 3D. Les images doivent ensuite être analysées pour faire correspondre des angles de rotation appliqués à l’objet 3D de départ, afin d’obtenir dans le microscope la projection observée. Une projection contient toute l’information structurale pour remonter aux coordonnées atomiques de l’objet 3D. En utilisant un grand nombre de données, on peut ainsi obtenir la structure tridimensionnelle d’un objet biologique à haute résolution. Les recherches qui prenaient plusieurs mois pour obtenir la structure d’un objet biologique ne prennent plus que quelques semaines, voire quelques jours.
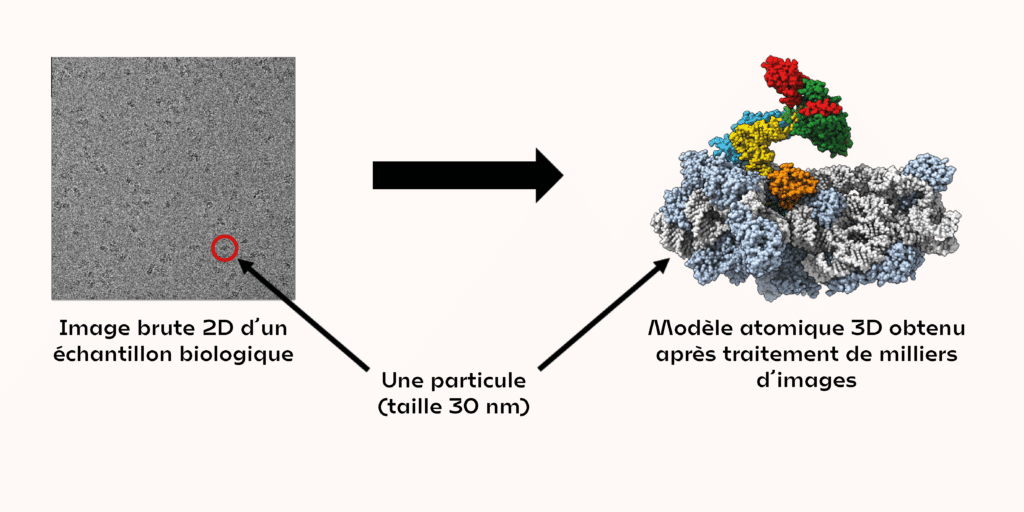
Tout échantillon biologique ne peut pas encore être observé avec un microscope électronique : à l’heure actuelle, les objets inférieurs à 20–25 kDa (environ 3 nm) sont très difficiles, voire impossible à observer. Mais c’est là que les autres approches structurales décrites au début peuvent combler ce manque !
Un des laboratoires de l’Ecole polytechnique (BIOC) est pionnier dans son domaine en utilisant l’approche par microscopie électronique pour étudier, parmi ses différents thèmes de recherche, les ribosomes d’Archées. Les données préliminaires aux travaux publiés910 ont été réalisés au CIMEX (voir encadré).
Encore une autre révolution à venir ?
Depuis une dizaine d’années, des révolutions majeures ont pu être observées dans la conception des microscopes, dans la technologie des caméras utilisées pour prendre les images et dans les logiciels utilisés pour traiter ces images. Cette discipline, mise au goût du jour, a changé radicalement l’approche qu’avaient les chercheurs pour répondre à un problème biologique.
La prochaine étape est d’aller voir les molécules dans leur contexte cellulaire : la tomographie électronique est encore une méthode artisanale qui demande encore beaucoup de savoir-faire et qui n’est pas très démocratisée. La préparation des échantillons est encore plus critique : il faut pouvoir découper dans un glaçon de cellule une fine lamelle d’environ 200 nm d’épaisseur qui contient la région d’intérêt. Cette lamelle est ensuite observée sous différents angles dans le microscope pour calculer une carte 3D de la région d’intérêt à moyenne résolution. Les ordinateurs sont capables d’aller reconnaître ensuite dans cette carte 3D des profils de molécules pour reconstruire un modèle de la carte 3D. Les développements dans cette discipline sont nombreux et devraient permettre dans quelques décennies de pouvoir regarder n’importe où dans la cellule une protéine particulière dans son contexte cellulaire. Le nano-univers des cellules n’aura bientôt plus de secret pour nous…
CIMEX
Le Centre Interdisciplinaire de Microscopie Electronique de l’École polytechnique (CIMEX) est une plateforme hébergeant plusieurs microscopes où physiciens, chimistes et biologistes peuvent se rencontrer et utiliser des microscopes électroniques performants. L’un des microscopes, appelé NanoMAX, est un prototype mondial. Il est utilisé par les physiciens et les chimistes et permet d’étudier, entre autres, à haute résolution et en temps réel la croissance des nanotubes de carbone. L’autre microscope plus utilisé par les biologistes, appelé Nan’eau, est un microscope polyvalent et bien équipé. Il permet d’obtenir des données préliminaires importantes avant de passer à des microscopes de pointe (disponibles sur 3 centres de référence au niveau national). Les images alors enregistrées seront de qualité suffisante pour obtenir la structure 3D à haute résolution de l’objet étudié.
















