
La biodiversité est en pleine crise1 et il est à la fois important de suivre l’évolution des populations sauvages et de les déranger le moins possible. Deux injonctions a priori contradictoires qu’il devient possible de concilier grâce à une source d’informations de plus en plus utilisée : l’ADN environnemental (ou ADNe).

Pister les organismes grâce à leur ADN
Dès les années 80, des échantillons collectés dans l’environnement ont permis d’étudier les micro-organismes invisibles qu’ils contenaient, via l’analyse de leurs génomes. Cette approche, développée grâce aux progrès des techniques de séquençage et des outils informatiques, a donné naissance à la métagénomique, c’est-à-dire l’étude à large échelle des génomes (à laquelle nous avons déjà consacré une chronique).
Mais s’il y a des micro-organismes un peu partout, ils ne sont pas les seules sources d’ADN dans l’environnement ! Mucus, peau mortes, poils, carcasses, déjections… Tous les êtres vivants, quelle que soit leur taille, laissent des traces de leur passage. Et celles-ci peuvent contenir de l’ADN permettant de suivre leur présence, ce qui a été démontré pour la première fois en 2008 par une équipe du Laboratoire d’écologie alpine, qui a identifié l’ADN de Grenouilles taureau dans différentes mares2.
L’ADNe est donc également un outil de suivi des populations de macro-organismes, dont l’efficacité a été démontrée dans de nombreux milieux, des sédiments aux fonds marins… Jusqu’à l’air lui-même ! Deux études publiées début 202234 ont prouvé qu’en aspirant de l’air dans des zoos et en analysant son contenu en ADN, il est possible d’identifier des dizaines d’espèces d’animaux vivant dans ou à proximité de ces zoos. Cette approche prometteuse ouvre de nouvelles possibilités mais a encore un certain nombre de faiblesses. Pour les comprendre, il faut se pencher plus en détail sur son fonctionnement.
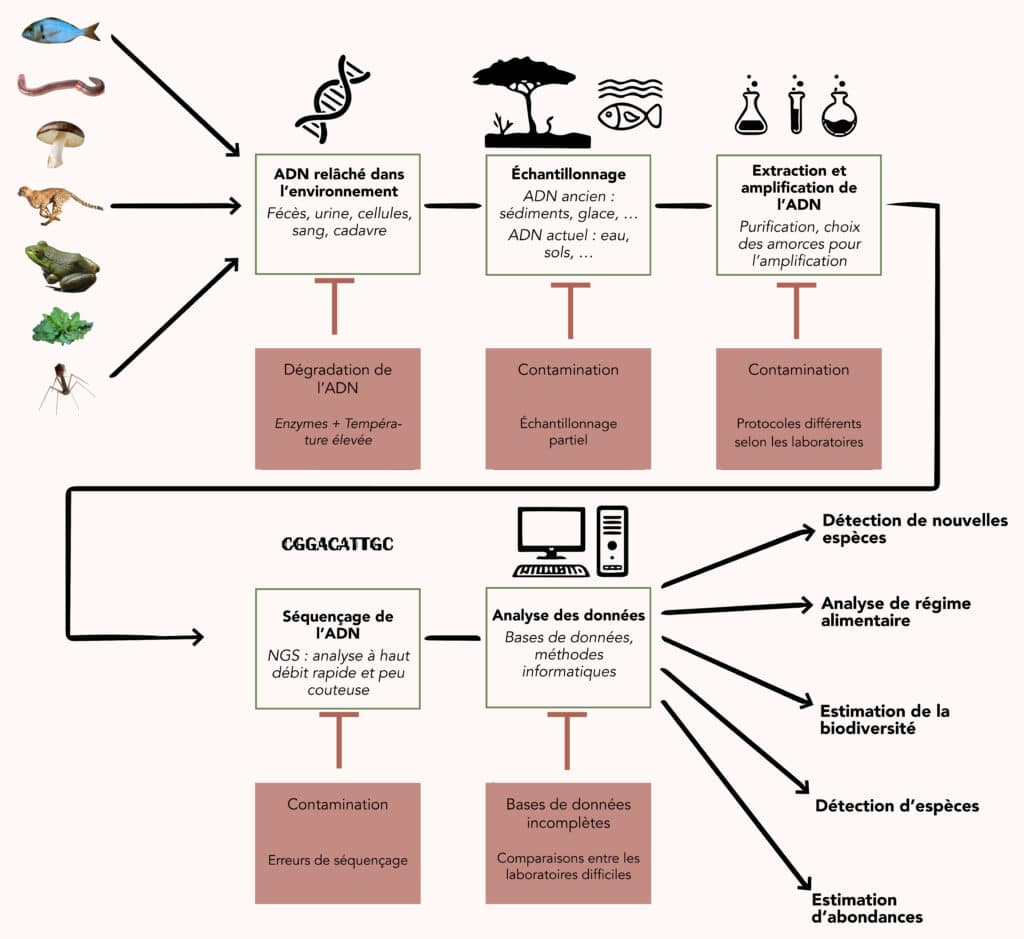
Faire parler l’ADN environnemental
Une fois récupéré dans l’environnement, l’ADN peut être analysé de trois façons. Une première consiste à faire de la métagénomique globale, en séquençant l’ensemble de l’ADN pour tenter d’identifier un maximum des génomes présents. Cette approche est adaptée à l’étude des micro-organismes, qui sont directement contenus dans l’échantillon et dont l’ADN est donc bien préservé et présent en grandes quantités. Elle est moins pertinente pour les macro-organismes, dont l’ADN est plus rare et plus abîmé dans l’environnement, se trouvant généralement sous la forme de fragments de quelques dizaines à quelques milliers de nucléotides de long.
L’analyse de ce type d’ADNe repose sur l’utilisation de codes-barres moléculaires (barcodes), des séquences génétiques permettant d’identifier une espèce ou une catégorie d’organismes. Celles-ci doivent être assez courtes pour être repérables dans de l’ADN naturellement fragmenté. Ces séquences « codes-barres » sont amplifiées spécifiquement par PCR puis séquencées et analysées. Elles peuvent être plus ou moins spécifiques, permettant de suivre une espèce unique, un ensemble d’espèces proches ou de répertorier plus largement la biodiversité d’un environnement. Selon l’amplitude choisie, on parle de « barcoding » ou de « métabarcoding », qui est une forme de métagénomique ciblée.
L’analyse repose ensuite sur la comparaison des codes-barres obtenus avec ceux répertoriés dans des bases de données. Celles-ci sont plus ou moins bien alimentées selon les domaines et c’est un des points faibles de l’étude de l’ADNe : plus les bases de données seront riches, moins les analyses seront limitées. L’accumulation de nouvelles données fait progressivement évoluer la situation !
Forces et faiblesses de l’ADN environnemental
À l’heure actuelle, l’ADNe a quatre types principaux d’applications : l’étude de la biodiversité (catalogage des espèces, suivi dans le temps, analyse des fonctions biologiques6 …) ; le suivi ciblé de certaines espèces (notamment des espèces menacées, invasives ou bioindicatrices7) ; l’estimation de l’abondance relative d’espèces dans un milieu donné et la reconstruction de régimes alimentaires en analysant l’ADN contenu dans des excréments8.
Dans certains milieux, comme les sédiments et les environnements très froids, l’ADNe est préservé pendant de longues périodes, ce qui permet de remonter le temps. Des chercheurs ont ainsi étudié des unicellulaires de la rade de Brest sur une période de 1 400 ans, faisant ressortir l’impact de la Seconde Guerre mondiale et des changements récents de pratiques agricoles9. Le record d’utilisation d’ADNe en paléoécologie a été repoussé début décembre grâce à des échantillons de pergélisol du Groenland, qui ont permis de reconstituer un paléo-écosystème d’environ deux millions d’années10 !
Dans certains milieux, comme les sédiments et les environnements très froids, l’ADNe est préservé pendant de longues périodes, ce qui permet de remonter le temps.
L’ADNe présente également de nombreux avantages pour l’étude des espèces actuelles. En effet, son prélèvement est non invasif, ce qui évite de perturber les milieux étudiés, et très simple. Le travail de terrain correspondant nécessite peu de matériel et de formation, permet d’accéder à des lieux inadaptés aux observations directes, peut être greffé facilement sur des expéditions déjà prévues par ailleurs et reste découplé du travail d’analyse. Beaucoup moins coûteux et contraignant que les méthodes d’observation classiques, il permet de multiplier les prélèvements et ouvre des possibilités de surveillance à large échelle spatiotemporelle. Les méthodes d’analyse se prêtent elles aussi à cet élargissement, car mutualiser le traitement de nombreux échantillons génère des économies d’échelle.
Aussi prometteuse soit-elle, l’utilisation de l’ADNe a cependant des limites. Pour commencer, et c’est fondamental même si cela paraît évident, détecter l’ADN d’un individu n’équivaut pas à détecter sa présence. Cela ne dit rien de son état de santé, de sa taille ou de son stade de développement, autant d’informations qui ne restent accessibles que par des observations directes. Cela n’informe pas forcément non plus sur sa localisation exacte, puisque l’ADN peut être transporté dans l’environnement ! Dans les cours d’eau, les espèces peuvent ainsi laisser des traces sur plusieurs kilomètres en aval de leur position réelle.
Par ailleurs, tous les organismes ne libèrent pas de l’ADN de façon comparable dans leur environnement et, selon la fragilité de la structure contenant l’ADN et les conditions du milieu (notamment de pH et de température), l’ADNe peut être dégradé plus ou moins rapidement. Absence d’ADN ne rime donc pas forcément avec absence d’une espèce. Àl’inverse, de l’ADN peut facilement venir contaminer des échantillons, qu’il vienne des expérimentateurs et de leur matériel ou d’une autre source. Des restaurants ou des marchés en zone côtière peuvent par exemple conduire à la détection d’ADNe de poissons ne vivant pas sur place12.
Enfin, les techniques de traitements biomoléculaires et informatiques génèrent leurs propres biais. Au-delà du caractère incomplet des bases de données, l’amplification par PCR n’est pas aussi efficace sur toutes les séquences d’ADN et, selon les codes-barres choisis et la façon de les détecter, des faux négatifs ou des faux positifs peuvent apparaître, ce qui est difficile à surveiller au cas par cas dans les analyses à grande échelle. Ajoutée aux biais d’échantillonnage, cette variabilité de l’amplification limite la pertinence de l’ADNe comme outil de quantification. Chaque étude de ce type nécessite des vérifications méticuleuses pour s’assurer que les résultats obtenus via ADNe sont comparables à ceux obtenus en comptage manuel. Dernière cerise sur le gâteau des complications : le séquençage lui-même peut être source d’erreur.
La résolution de ces problèmes techniques est un enjeu central pour les équipes s’intéressant à l’ADNe. Plusieurs projets de recherche visent ainsi à réduire les incertitudes d’analyse, notamment en standardisant les protocoles, en alimentant les bases de données et en répertoriant des codes-barres moléculaires pertinents13, ce qui présage des améliorations à venir.
Pour reprendre la formulation utilisée par Sam Chew Chin, doctorant étudiant les populations de poissons via l’ADNe, cet outil peut être considéré comme un « nez génétique », une « nouvelle façon de sentir la biosphère ». Il ne permet pas de tout détecter de façon parfaite, mais il ouvre des possibilités qui, combinées aux autres approches, ne pourront qu’améliorer notre compréhension de la biodiversité.








